Genome Biology:新型工具,有效实现了批次效应的去除和空间坐标的对齐
时间:2023-11-01 17:41:54 热度:37.1℃ 作者:网络
单细胞组学技术的发展有利于解析组织器官中复杂的细胞组成,为研究癌细胞的异质性,探索癌症不同亚型的临床治疗提供分子学基础。然而,单细胞技术损失了细胞在组织中的空间位置信息,无法探索在重要的生命过程中细胞与细胞之间的相互作用机制。近年来,基于测序和基于成像的空间转录组技术得到了迅速发展,为解析细胞的空间分布和功能提供了新的可能。
大量空间转录组数据的出现,可以为人类细胞图谱(HCA)构建通用坐标系(common coordinate framework),从而在特征空间和物理空间中实现细胞的整合、查询和定位。为整合不同来源的空间转录组数据,需要解决两个主要挑战:1)消除批次效应;2)对齐不同切片的空间坐标。在单细胞转录组上广泛使用的批次效应去除方法,旨在去除不同批次之间有相同/相似细胞类型之间的表达差异,并未考虑空间转录组数据的独特特征(如细胞基因表达的空间依赖性和空间一致性等)。因此会导致批次矫正不充分,同时矫正后的数据无法保留其空间特性,进而影响空间域的划分和空间基因的查找。
为此,北京交通大学李翔宇、美国德州大学达拉斯分校张奇伟课题组联合开发了一个整合不同切片、不同实验条件、不同实验技术的空间转录组样本的新工具SPIRAL。此工具有效地实现了批次效应的去除和空间坐标的对齐。研究成果以“SPIRAL: integrating and aligning spatially resolved transcriptomics data across different experiments, conditions, and technologies”为题,于2023年10月20日发表于Genome Biology期刊上。
该工具包含两个模块SPIRAL-integration和SPIRAL-alignment。SPIRAL-integration通过整合归纳式图神经网络结构GraphSAGE、解耦网络和解码器来学习细胞的低维表示。它旨在保留特征空间相似性和物理空间相似性的同时,对低维表示进行噪音部分和生物部分的解耦学习。解耦网络分别包含两部分:生物判别器和噪音分类器,生物判别器通过使用参数化的梯度反转策略来学习与样本批次信息无关的生物表示,而噪音分类器则专注学习样本特异的批次表示。SPIRAL-integration可以得到去除批次效应后的低维表示和基因表达,实现了多样本联合空间域划分和基因表达的降噪。以SPIRAL-integration所得到的空间域划分为基础,SPIRAL-alignment模块以空间域为单位,采用Gromov-Wasserstein 距离实现空间坐标对齐,使得相同空间域的细胞或spot具有相似的空间坐标。(图1)
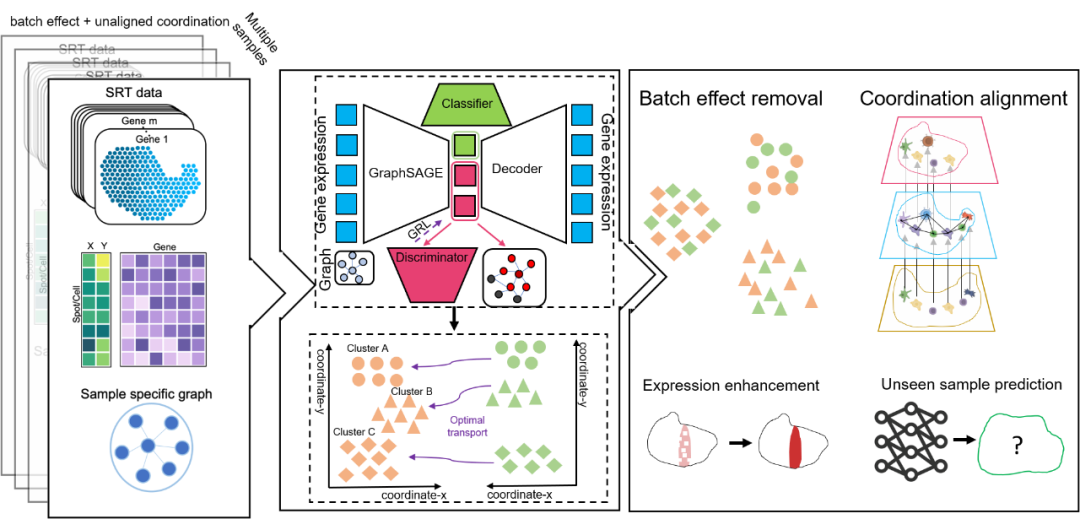
图1 SPIRAL算法工作流程图
研究人员在不同样本、不同实验条件、不同实验技术产生的空间转录组数据集上验证了SPIRAL的优越性能与稳定性。与其他方法相比,SPIRAL在消除批次效应、确保多样本空间域划分的一致性和准确性上具有卓越表现。此外,SPIRAL具备以下功能:1)提升低分辨率数据的精度;2)预测新的空间转录组数据的空间域并进行空间坐标检索;3)对新的单细胞转录组数据实现细胞类型迁移;4)基因表达的降噪。
在已标注好的10X Visium技术产生的人类背外侧前额叶皮质数据上,SPIRAL实现了比现有方法更好的批次效应去除和多样本的空间域划分;在10X Visium技术生成的小鼠大脑的冠状面数据上,SPIRAL成功划分了前后脑连接的空间域,并精确识别出小脑中更加细致的颗粒细胞层(granule layer)和浦肯野细胞层(purkinje cell layer);针对三种不同样本处理方式的小鼠大脑矢状面的样本数据(新鲜样本+H&E染色、新鲜样本+DAPI染色、石蜡包埋样本+H&E染色),SPIRAL相较于其他方法,更为高效地去除了批次效应,并成功识别出这三种样本共有的海马区的精细结构。此外,SPIRAL还能比较由不同实验技术产生的数据,并有效地进行空间域迁移(图2)。在多个数据集中,SPIRAL的空间坐标对齐能力都优于PASTE。
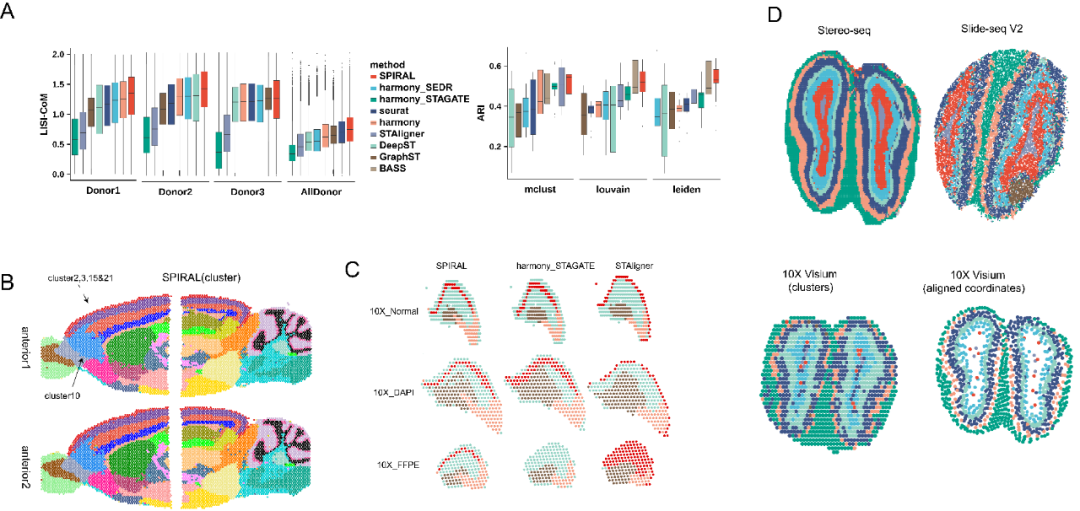
图2 多个数据集的部分结果
综上所述,该研究提出了整合多样本空间转录组的方法SPIRAL,可以有效实现对多样本、多实验流程、多实验技术的样本的批次效应去除和空间坐标对齐。随着空间转录组技术的不断发展以及大规模数据集的不断产生,SPIRAL有望成为空间转录组数据整合的有力工具,为构建人类细胞图谱和通用空间坐标提供助力。


