【协和医学杂志】Notch信号通路与先天性心脏病
时间:2024-07-31 15:00:14 热度:37.1℃ 作者:网络
综 述
先天性心脏病(CHD)是最常见的人类发育出生缺陷,指胎儿在胚胎时期的心脏发育异常,通常伴有大血管发育不良,其发病机制复杂,目前尚未阐明。Notch信号通路最早于1917年在果蝇体内发现,其广泛存在于无脊椎动物及脊椎动物中,在各物种进化过程中高度保守[1]。该通路在哺乳动物心脏的房室管(AVC)、流出道(OFT)、主动脉瓣及心室发育过程中均起到重要的调控作用,可促进心肌细胞再生、参与心脏血管构建,并通过负性调节心肌成纤维细胞向肌成纤维细胞转化修复心肌。研究表明,Notch信号通路的异常及Notch基因突变会导致CHD的发生。本文就Notch信号通路参与心脏发育及导致CHD的作用机制展开综述,以期为CHD的早期诊断提供参考。
1 Notch信号通路
Notch信号通路由Notch受体(Notch 1~4),Notch配体(JAG1~2、DLL1~4),细胞内效应器分子[包括CSL(CBF-1/Suppressor of hairless/ Lag-1) DNA结合蛋白、毛发分裂增强因子(Hes)基因家族、Hey(hairy and enhancer-of-split related with YRPW motif)基因]组成[2-3];Notch活化过程主要以经典的CBF-1/RBP-JK(C-promoter binding factor 1/recombination signal sequence-binding protein J)依赖途径为主,详见图1。
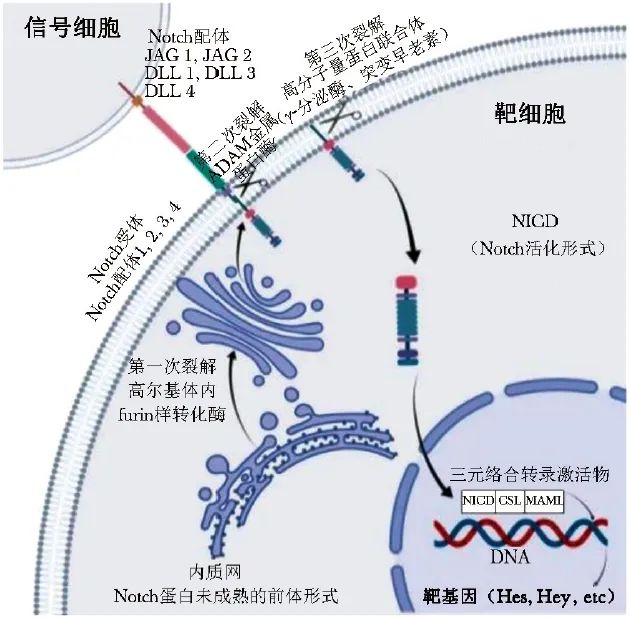
图1 Notch信号通路活化经典途径
目前,哺乳动物中存在4种Notch基因(Notch 1~4),其中Notch 1~3与人类疾病有关[4]。动物实验表明,Notch 1在哺乳动物胚胎时期心血管系统的发育过程中发挥重要作用,如在大鼠的胚胎发育阶段,心脏发育最初部位中胚层中即可检测到Notch 1的表达[5]。同时,Notch 1突变与钙化性主动脉瓣膜病(CAVD)发生率成正相关[6]。Notch 2突变则会导致Alagille综合征(ALGS)[7]。而Notch 3与动脉平滑肌细胞发育有关,其突变与显性遗传病有关。
2 Notch信号通路参与心脏发育
Notch在哺乳动物线性心管形成前后的表达特性已被证实。动物实验表明,在大鼠胚胎期第7.5天,Notch 1首先表达于中胚层,第8天后局限于原始心管OFT内膜层,从而调控心脏的形态,Notch 4表达于心内膜,至胚胎期第12.5天可在心室OFT房室区、心房肌和心室肌小梁中检测到JAG1的表达,而Notch 2直到胚胎期第13.5天才出现在心房和心室肌中[8]。Notch信号通路可参与房室管、心脏传导系统、心室、OFT、冠状动脉的发育。
2.1 房室管形成
在AVC的形成过程中,部分心内膜细胞在临近心肌信号刺激下发生改变,Notch信号通路活化促进细胞上皮——间质转化 (EMT)形成腹侧、背侧两个心内膜垫,二者相对生长、彼此融合将房室总管分隔,同时,心内膜垫也参与瓣膜组织的形成[9]。Abu等[10]研究发现,斑马鱼瓣膜组织发育的基因表达与哺乳动物相似,JAG 1b、Hey1、Hey2基因参与编码AVC心内膜关键成分。因此Notch信号通路的活化是诱导EMT的必要条件,促进房室结的形成。
骨形态发生蛋白2(BMP2)是AVC分化和EMT发生的关键信号,BMP2失活则会破坏EMT[11]。Andrés-Delgado等[12]研究发现,心肌中Notch 1异位激活导致非腔室心肌表达Hey1,从而抑制BMP2的表达并破坏瓣膜EMT,而心内膜细胞中Notch 1可上调AVC心肌中BMP2的表达,二者呈正相关。
此外,Notch信号通路靶基因Hey1通过抑制BMP2在房室中的表达,将其和Tbx 2的表达限制于AVC中,进而促进AVC的成熟,影响房室总管的分隔以及房室瓣的形成[13]。因此,Notch信号通路在心脏发育的不同部位对BMP2的调控作用有所差异,可将BMP2及促AVC形成相关基因限制于AVC中,以促进AVC的发育。
2.2 心脏传导系统发育
在心脏起搏点窦房结(SAN)形成过程中,Notch 1信号在SAN的心内膜中募集。Wang等[14]研究发现,在心脏发育过程中,静脉窦瓣(SVV)与SAN共同发育,而在Notch 1基因敲除的胚胎心肌SVV中Wnt2基因表达明显降低,说明Wnt2可通过Wnt/β-catenin信号通路参与SVV的形成。因此,Notch 1可上调Wnt信号促进SVV与SAN的形成。
2.3 心室发育
早期心室发育首先是心肌小梁的形成,而Notch 1存在于小梁底部的心室心内膜细胞中,其活性可影响BMP10的表达,进而调控心肌细胞增殖。
此外,Notch信号通路还影响激活心肌小梁形成的EFNB2通路和神经调节素1(NRG1)-ERBB通路,心内膜Notch通路通过作用于神经嵴衍生物表达蛋白2(HAND2)调控NRG1的表达,而EFNB2是NRG1的上游因子,在免疫球蛋白Kappa J区重组信号结合蛋白(RBPJ)突变体中外源性给予NRG1可恢复心肌细胞分化缺陷[13],说明NRG1可促进心肌小梁形成[15];
另有研究发现,早期心室发育中的DLL4向β-1,3-N-乙酰葡糖胺基转移酶Manic Fringe(β-1,3-N-acetylglucosaminyltransfe-rase manic fringe,MFNG)重组蛋白修饰的Notch 1受体发出信号并促进心肌压紧[13]。动物实验表明,小鼠胚胎心肌细胞表达的Hey2可促进致密心肌细胞向小梁层扩张,进而促进内小梁和外致密层之间心肌杂交带的形成以及心室压实[16]。
综上,Notch信号通路可通过调控BMP10、EFNB2通路、NRG1-ERBB通路中相关蛋白表达以及上调下游基因促进早期心室发育。
2.4 OFT发育
Notch信号通路在第二心野(SHF)衍生的心脏结构中发挥关键作用。SHF是指心脏发育所需心脏祖细胞的第二来源,其对心肌、心内膜以及间充质细胞均有影响,而这些细胞可形成OFT结构。Notch 1可下调SHF阳性细胞Wnt/β-catenin信号传导以促进SHF中的心脏祖细胞分化[13]。
De Zoysa等[17]研究发现,在小鼠心脏发育早期敲除DLL4后会导致SHF细胞凋亡率上升4倍以及OFT发育不足。Rammah等[18]发现,Notch配体、Notch 1以及非经典的配体DLK1 (delta-like noncanonical Notch ligand 1)通路可负调控过氧化物酶体增殖物激活受体γ(PPARγ),从而保证OFT正常发育。因此,Notch信号通路可通过促进Wnt信号通路及DLL4促进SHF分化,同时其配体及受体可直接促进OFT发育。
2.5 冠状动脉发育
Notch信号通路与心脏冠状动脉发育有关,参与冠脉血管壁的形成。血管内皮生长因子(VEGF)在血管形成中发挥重要作用,其通过结合血管内皮细胞生长因子受体(VEGFR-2)促进血管出芽,而Notch受体可抑制VEGFR-2的表达,Notch配体DLL4可抑制血管新生芽的形成[19]。
Travisano等[20]研究发现,Notch配体JAG1和DLL4拮抗调节冠状动脉初级动脉丛的形成,且EphrinB2是拮抗过程中的关键效应因子,随后的冠状动脉分化依赖于DLL4-JAG1-EphrinB2信号级联反应。
此外,Notch信号通路通过调节心外膜细胞向上皮细胞-间充质的转化影响冠状动脉平滑肌细胞分化,其作用于转化生长因子-β(TGF-β)的上游信号[21],TGF-β可与Notch信号通路共同调控冠状动脉的正常发育[22]。
McCallinhart等[23]首次发现,Notch配体JAG1及其受体可在冠状动脉肌内皮连接(MEJ)内表达,平滑肌可借助于该连接,接受血液或内皮细胞的化学信息。因此,Notch信号通路可通过双重调节VEGF来调控血管出芽,其与配体结合通过EphrinB2拮抗调节冠状动脉初级动脉丛的形成,促进冠状动脉分化,同时也可作用于TGF-β上游信号促进冠状动脉形成。
3 Notch信号通路与CHD
3.1 CHD概述
CHD是先天性疾病中较常见的一类。根据《常见先天性心脏病经皮介入治疗指南(2021版) 》,CHD包括室间隔缺损(VSD)、房间隔缺损(ASD)、动脉导管未闭(PDA)、肺动脉狭窄(PS)、法洛四联症(TOF)、左心发育不全综合征(HLHS)等[24]。全球新生儿中CHD患病以VSD、ASD为主[25]。上述研究表明,Notch信号通路能够作用于心脏发育各个阶段,Notch基因突变与CHD发病密切相关。
3.2 Notch信号通路异常导致CHD
3.2.1 HLHS
HLHS是以左心室、二尖瓣、主动脉瓣和升主动脉左心部分发育不良为特征的严重单心室CHD。研究表明,Notch靶基因启动子位点赖氨酸甲基转移酶2D(KMT2D)缺陷会导致HLHS患者冠状动脉畸形[26]。此外,Notch信号通路异常会导致瓣膜形成异常[27],SHF祖细胞中HAND2和Hey2基因缺失分别导致三尖瓣闭锁(TA)和VSD[28]。如阻断心内膜Notch-RBPJ通路会造成RBPJ表达缺失及VSD[29]。因此,Notch下游靶基因缺陷导致冠状动脉畸形从而引起左心部分发育不良,此外Notch信号通路异常使下游信号缺失可导致瓣膜形成异常以及VSD。
3.2.2 TOF
TOF是具有PS、VSD、主动脉骑跨以及右心室肥大4种异常表现的紫绀型先天性心脏畸形。Page等[30]发现,Notch 1基因位点变异会引发非典型性TOF。研究表明,TOF患者右心室OFT蛋白δ样DLK1的表达显著低于对照组,DLK1启动子低甲基化从而抑制DLK1基因的转录活性导致TOF[31],提示Notch 1基因位点变异以及Notch配体基因转录活性下降均会导致TOF。
3.2.3 PDA
PDA是婴儿出生后动脉导管持续未闭保持开放的病理状态,常合并血管畸形,严重时可表现为艾森曼格综合征。Ovali等[32]研究发现,Notch受体对小鼠收缩性平滑肌细胞分化发挥正性作用。此外,JAG1-Notch信号轴也存在于平滑肌细胞中,在血管发育期间可促进平滑肌细胞分化及导管的解剖闭合[33]。因此,Notch受体与配体均可正性调节血管平滑肌细胞分化,其缺失会导致有特定作用的细胞相关基因表达下调,从而影响动脉导管解剖重塑,进而导致PDA。
3.2.4 二叶式主动脉瓣病变
二叶式主动脉瓣病变(BAV)在CHD中的发病率仅为1%~2%,目前相关研究较少,病因尚不明确。Kostina等[34]研究发现,左心室OFT畸形患者中Notch 1基因突变与BAV相关,BAV患者主动脉内皮细胞主动下调Notch信号,且所有Notch配体均无法上调EMT,导致Notch信号通路异常,从而引起内皮功能障碍及主动脉壁改变。
在BAV患者升主动脉活检过程中发现, Sirtuin 1 (SirT1)的激活与Notch信号通路之间存在负反馈调节[35]。对SirT1敲除小鼠进行研究发现,该小鼠在产前或围产期会死于心脏瓣膜畸形,表明SirT1参与心脏发育与瓣膜形成[36]。综上,这些分子或可作为潜在生物标志物,为BAV的早期诊断提供新思路。
3.2.5 CAVD
Ackah 等[37]研究发现,在非综合征型常染色体显性遗传的人类家系中,Notch 1突变会引起一系列发育性主动脉瓣异常和严重的瓣膜钙化,Notch 1的转录本在正在发育的小鼠主动脉瓣中表现活跃,Notch 1信号通路激活的毛发相关转录抑制因子(Hrt)家族与转录因子Runx2相互作用,抑制Runx2转录活性,而Runx2与主动脉瓣钙化密切相关,是成骨细胞的核心转录调节因子。
另有研究表明,Notch 1突变可导致钙盐沉积的去抑制,进而导致主动脉瓣钙化[38]。Acharya等[39]对离体猪主动脉瓣钙化模型进行研究发现,抑制Notch信号活性则钙化加速,同时主动下调Notch信号后,转录因子Sox9及其靶基因表达显著下调,反之,刺激Notch信号则钙化减慢,说明Notch信号通路异常可通过不同途径导致主动脉瓣钙化。
3.2.6 ALGS
研究表明,JAG1和Notch 2突变会导致ALGS的发生,且大多数Notch 2突变是错义突变[40]。Gilbert等[41]研究发现,ALGS患者中约94.5%存在JAG1突变(包括错义突变、无义突变、剪接突变等),2.5%的患者存在Notch 2突变,为临床诊断提供了新思路。动物实验表明,小鼠JAG1纯合突变为致死性突变,而JAG1杂合小鼠仅表现为眼部异常,无心脏异常表现,Notch 2单倍剂量不足的小鼠则表现为ALGS特征性多系统病变[42],这表明Notch 2及JAG1对心脏发育均具有重要意义。
4 小结与展望
综上所述,Notch信号通路在心脏生长发育时期发挥重要调节作用,影响心脏的形态结构及正常功能。该通路异常可通过介导HAND2、Runx2等因子的缺失以及JAG、DLK1等的异常表达导致CHD,未来或可进一步对这一通路的下游信号分子进行检测,从而提高胎儿CHD的检出率。
参考文献
[1]Zhou B H, Lin W L, Long Y L, et al. Notch signaling pathway: architecture, disease, and therapeutics[J]. Signal Transduct Target Ther, 2022, 7(1): 95.
[2]王雪淞, 周林, 李林材, 等. Notch信号通路调控间充质干细胞的增殖与分化[J]. 中国组织工程研究, 2024, 28(19): 3076-3083.
[3]李婷, 程亚楠, 周小平. 阻断Notch信号通路后补益营卫方对衰老皮肤表皮干细胞Notch1、Jagged1、RBP-Jκ和Hes1表达的影响[J]. 中国老年学杂志, 2023, 43(4): 876-880.
[4]张岩, 谢新明, 韩冬, 等. Notch信号通路与心血管疾病的研究进展[J]. 医学综述, 2016, 22(14): 2748-2750.
[5]Buijtendijk M F J, Barnett P, Van Den Hoff M J B. Development of the human heart[J]. Am J Med Genet C Semin Med Genet, 2020, 184(1): 7-22.
[6]Kraler S, Blaser M C, Aikawa E, et al. Calcific aortic valve disease: from molecular and cellular mechanisms to medical therapy[J]. Eur Heart J, 2022, 43(7): 683-697.
[7]Yang Q, Wu F, Mi Y P, et al. Aberrant expression of miR-29b-3p influences heart development and cardiomyocyte proliferation by targeting NOTCH2[J]. Cell Prolif, 2020, 53(3): e12764.
[8]Langa P, Shafaattalab S, Goldspink P H, et al. A perspective on Notch signalling in progression and arrhythmogenesis in familial hypertrophic and dilated cardiomyopathies[J]. Philos Trans R Soc Lond B Biol Sci, 2023, 378(1879): 20220176.
[9]Poelmann R E, Gittenberger-De Groot A C. Hemodynamics in cardiac development[J]. J Cardiovasc Dev Dis, 2018, 5(4): 54.
[10]Abu Nahia K, Migda M, Quinn T A, et al. Genomic and physiological analyses of the zebrafish atrioventricular canal reveal molecular building blocks of the secondary pacemaker region[J]. Cell Mol Life Sci, 2021, 78(19/20): 6669-6687.
[11]Prados B, Gómez-Apiñániz P, Papoutsi T, et al. Myocardial Bmp2 gain causes ectopic EMT and promotes cardiomyocyte proliferation and immaturity[J]. Cell Death Dis, 2018, 9(3): 399.
[12]Andrés-Delgado L, Galardi-Castilla M, Münch J, et al. Notch and Bmp signaling pathways act coordinately during the formation of the proepicardium[J]. Dev Dyn, 2020, 249(12): 1455-1469.
[13]MacGrogan D, Münch J, De La Pompa J L. Notch and interacting signalling pathways in cardiac development, disease, and regeneration[J]. Nat Rev Cardiol, 2018, 15(11): 685-704.
[14]Wang Y D, Lu P F, Jiang L P, et al. Control of sinus venous valve and sinoatrial node development by endocardial NOTCH1[J]. Cardiovasc Res, 2020, 116(8): 1473-1486.
[15]Ye S Q, Wang C K, Xu Z H, et al. Impaired human cardiac cell development due to NOTCH1 deficiency[J]. Circ Res, 2023, 132(2): 187-204.
[16]Tsedeke A T, Allanki S, Gentile A, et al. Cardiomyocyte heterogeneity during zebrafish development and regeneration[J]. Dev Biol, 2021, 476: 259-271.
[17]De Zoysa P, Toubat O, Harvey D, et al. Murine model of cardiac defects observed in Adams-Oliver syndrome driven by Delta-like ligand-4 haploinsufficiency[J]. Stem Cells Dev, 2021, 30(12): 611-621.
[18]Rammah M, Théveniau-Ruissy M, Sturny R, et al. PPARγ and NOTCH regulate regional identity in the murine cardiac outflow tract[J]. Circ Res, 2022, 131(10): 842-858.
[19]Neffeová K, Olejnícˇková V, Naňka O, et al. Development and diseases of the coronary microvasculature and its communication with the myocardium[J]. WIREs Mech Dis, 2022, 14(5): e1560.
[20]Travisano S I, Oliveira V L, Prados B, et al. Coronary arterial development is regulated by a Dll4-Jag1-EphrinB2 signaling cascade[J]. Elife, 2019, 8: e49977.
[21]Tomanek R, Angelini P. Embryology of coronary arteries and anatomy/pathophysiology of coronary anomalies. A comprehensive update[J]. Int J Cardiol, 2019, 281: 28-34.
[22]Zhang Q, Wang L, Wang S Q, et al. Signaling pathways and targeted therapy for myocardial infarction[J]. Signal Transduct Target Ther, 2022, 7(1): 78.
[23]McCallinhart P E, Biwer L A, Clark O E, et al. Myoendothelial junctions of mature coronary vessels express notch signaling proteins[J]. Front Physiol, 2020, 11: 29.
[24]国家卫生健康委员会国家结构性心脏病介入质量控制中心, 国家心血管病中心结构性心脏病介入质量控制中心, 中华医学会心血管病学分会先心病经皮介入治疗指南工作组, 等. 常见先天性心脏病经皮介入治疗指南 (2021版)[J]. 中华医学杂志, 2021, 101(38): 3054-3076.
[25]艾珊珊, 何爱彬. 先天性心脏病基础研究进展[J]. 协和医学杂志, 2021, 12(3): 291-297.
[26]Yu Z Y, Zhou X, Liu Z Y, et al. KMT2D-NOTCH mediates coronary abnormalities in hypoplastic left heart syndrome[J]. Circ Res, 2022, 131(3): 280-282.
[27]Miao Y F, Tian L, Martin M, et al. Intrinsic endocardial defects contribute to hypoplastic left heart syndrome[J]. Cell Stem Cell, 2020, 27(4): 574-589.e8.
[28]Yu Z Y, Pek N M Q, Gu M X. Delving into the molecular world of single ventricle congenital heart disease[J]. Curr Cardiol Rep, 2022, 24(5): 463-471.
[29]Salguero-Jiménez A, Grego-Bessa J, D'Amato G, et al. Myocardial Notch1-Rbpj deletion does not affect NOTCH signaling, heart development or function[J]. PLoS One, 2018, 13(12): e0203100.
[30]Page D J, Miossec M J, Williams S G, et al. Whole exome sequencing reveals the major genetic contributors to nonsyndromic Tetralogy of Fallot[J]. Circ Res, 2019, 124(4): 553-563.
[31]Tian G X, He L L, Gu R Y, et al. CpG site hypomethylation at ETS1 binding region regulates DLK1 expression in Chinese patients with Tetralogy of Fallot[J]. Mol Med Rep, 2022, 25(3): 93.
[32]Ovali F. Molecular and mechanical mechanisms regulating ductus arteriosus closure in preterm infants[J]. Front Pediatr, 2020, 8: 516.
[33]Salvador J, Hernandez G E, Ma F Y, et al. Transcriptional evaluation of the ductus arteriosus at the single-cell level uncovers a requirement for vim (vimentin) for complete closure[J]. Arterioscler Thromb Vasc Biol, 2022, 42(6): 732-742.
[34]Kostina A S, Uspensky V E, Irtyuga O B, et al. Notch-dependent EMT is attenuated in patients with aortic aneurysm and bicuspid aortic valve[J]. Biochim Biophys Acta, 2016, 1862(4): 733-740.
[35]Abudupataer M, Zhu S C, Yan S Q, et al. Aorta smooth muscle-on-a-chip reveals impaired mitochondrial dynamics as a therapeutic target for aortic aneurysm in bicuspid aortic valve disease[J]. Elife, 2021, 10: e69310.
[36]Podyacheva E, Toropova Y. SIRT1 activation and its effect on intercalated disc proteins as a way to reduce doxorubicin cardiotoxicity[J]. Front Pharmacol, 2022, 13: 1035387.
[37]Ackah R L, Yasuhara J, Garg V. Genetics of aortic valve disease[J]. Curr Opin Cardiol, 2023, 38(3): 169-178.
[38]Garg V, Muth A N, Ransom J F, et al. Mutations in NOTCH1 cause aortic valve disease[J]. Nature, 2005, 437(7056): 270-274.
[39]Acharya A, Hans C P, Koenig S N, et al. Inhibitory role of Notch1 in calcific aortic valve disease[J]. PLoS One, 2011, 6(11): e27743.
[40]Kohut T J, Gilbert M A, Loomes K M. Alagille syndrome: a focused review on clinical features, genetics, and treatment[J]. Semin Liver Dis, 2021, 41(4): 525-537.
[41]Gilbert M A, Bauer R C, Rajagopalan R, et al. Alagille syndrome mutation update: comprehensive overview of JAG1 and NOTCH2 mutation frequencies and insight into missense variant classification[J]. Hum Mutat, 2019, 40(12): 2197-2220.
[42]Meester J A N, Verstraeten A, Alaerts M, et al. Overlapp-ing but distinct roles for NOTCH receptors in human cardiovascular disease[J]. Clin Genet, 2019, 95(1): 85-94.


