Nature Genetics:高通量空间转录组技术推动器官发生研究:MAGIC-seq为临床多组学研究提供新工具
时间:2024-09-13 06:03:24 热度:37.1℃ 作者:网络
引言
近年来,空间转录组学(Spatial Transcriptomics, ST)技术在揭示复杂组织的分子结构方面取得了显著进展,尤其是对胚胎发育、器官发生和肿瘤微环境等关键生物过程的研究具有重要意义。ST技术能够同时捕获组织的空间和分子特征,提供了前所未有的三维(3D)转录组图谱。然而,现有的ST方法在高效处理大规模样本、降低成本以及保持高通量的同时,确保大视野和3D体积兼容性方面仍存在诸多挑战。
9月10日Nature Genetics的研究报道“Custom microfluidic chip design enables cost-effective three-dimensional spatiotemporal transcriptomics with a wide field of view”,介绍了一种创新的微流体技术辅助的网格芯片用于空间转录组测序(MAGIC-seq)。该方法结合了碳二亚胺化学(Carbodiimide Chemistry)、空间组合索引(Spatial Combinatorial Indexing)以及微流体设计,显著提升了条形码的标记效率,并能够在降低成本的同时,实现对多种组织类型的敏感、稳定且可重复的检测。与传统的ST方法相比,MAGIC-seq通过增强微流体条形码标记效率,显著提高了通量,并且减少了批次效应。MAGIC-seq能够分析包括后生小鼠脑切片在内的复杂组织,达到接近单细胞分辨率的转录组解析,揭示了小鼠器官发生过程中的转录变化和动态轨迹。
MAGIC-seq的独特之处在于它能够高效、低成本地处理大视野的组织样本,同时保证了高分辨率。该方法生成的3D小鼠发育脑转录组图谱,包含93个切片,全面描绘了小鼠发育过程中的分子与细胞景观,为神经科学和发育生物学提供了宝贵的资源。这种高通量、低成本和大视野兼具的技术为未来的空间转录组研究开辟了新的途径。

随着转录组学技术的不断进步,研究人员可以更深入地了解复杂组织的分子结构,揭示细胞之间的相互作用及其在组织中的空间分布。空间转录组学(Spatial Transcriptomics, ST)是这一领域的前沿技术,它不仅能够分析组织的分子特征,还可以保持其空间信息。这种技术在器官发生、胚胎发育和肿瘤微环境的研究中发挥了至关重要的作用。然而,目前的ST技术面临着一些限制,如高成本、低通量以及难以处理大规模样本和三维(3D)组织体积。这些问题使得现有的ST方法在满足高效、低成本和大视野要求的同时难以兼顾。
为了应对这些挑战,研究团队开发了一种名为MAGIC-seq的创新技术。MAGIC-seq结合了碳二亚胺化学(Carbodiimide Chemistry)、空间组合索引(Spatial Combinatorial Indexing)和微流体设计,不仅提高了条形码标记效率,还大幅降低了实验成本。通过这一技术,研究人员能够对复杂组织进行高分辨率、低成本和高通量的空间转录组分析。
MAGIC-seq的核心技术包括三个主要步骤
定制微流体芯片设计:根据不同样本的需求,研究团队设计了多种芯片架构,包括三网格芯片和九网格芯片,这些芯片可以处理不同大小的样本,同时也能通过拼接网格芯片设计来扩大视野。例如,三网格芯片的每个区域为7 mm × 7 mm,包含4900个采集点(50 μm × 50 μm)。
空间条形码DNA阵列的预制:使用碳二亚胺化学反应将条形码X通过共价键固定到玻片上,然后使用第二个微流体芯片垂直排列条形码Y,与条形码X结合,从而生成一个空间组合索引的DNA阵列。此步骤极大地提高了条形码编码的效率,使得每个采集点都能拥有唯一的空间索引。
样本制备和测序:组织切片被固定到预先标记的玻片上,经过H&E染色、渗透处理后,mRNA被释放并被条形码捕获。接下来通过原位逆转录和cDNA扩增,生成包含空间条形码和cDNA插入物的文库,随后进行双端测序。通过这种方式,不仅可以获得组织的基因表达信息,还能保留其空间分布信息。
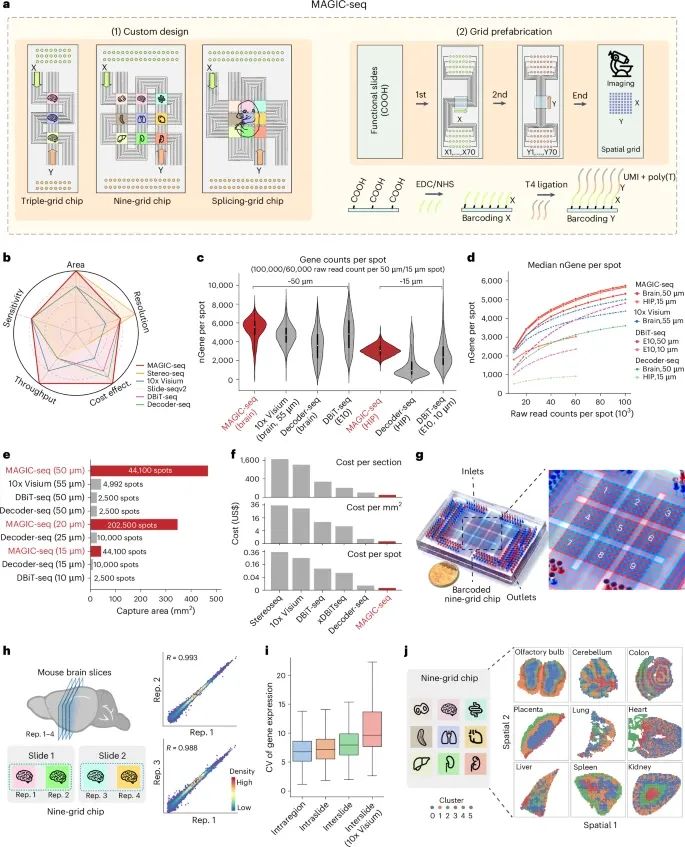
MAGIC-seq在多种组织类型中的高灵敏度、高通量和一致性分析结果(Credit: Nature Genetics)
MAGIC-seq的工作流程:图a概述了MAGIC-seq的主要工作流程,展示了不同的微流体芯片设计(如三网格芯片、九网格芯片和拼接网格芯片),这些芯片可以用于不同样本的处理。同时展示了如何通过空间条形码DNA阵列来捕获基因表达信息。
与其他空间转录组方法的比较:图b对MAGIC-seq与其他空间转录组方法(如DBiT-seq、Decoder-seq和10x Visium)的性能进行了比较,MAGIC-seq在灵敏度、通量、采集区域和成本方面表现出明显优势。
基因检测数量的对比:图c展示了MAGIC-seq相比其他方法在50 μm和15 μm分辨率下每个采集点检测到的基因数量,MAGIC-seq在这两个分辨率下均表现出更高的基因检测数量。
测序深度和基因数量的饱和曲线:图d展示了不同空间转录组方法在不同测序深度下检测到的基因数量饱和曲线,MAGIC-seq在同等测序深度下检测到的基因数量明显高于其他方法,显示出其更高的测序效率。
采集面积和成本的比较:图e和图f展示了MAGIC-seq与其他方法在采集面积、采集点数量和每个采集点的成本方面的比较,MAGIC-seq的采集面积更大,采集点数量更多,且每平方毫米的成本显著低于其他方法。
一致性和批次效应分析:图h和图i评估了MAGIC-seq在不同组织样本中的基因表达一致性,结果显示,MAGIC-seq在同一张玻片的不同切片之间以及不同玻片之间表现出极高的基因表达相关性,说明其具有较低的批次效应。
多组织类型的空间分布分析:图j展示了MAGIC-seq在九种不同小鼠组织中的空间基因表达分布,能够清晰区分组织的不同区域,显示了其在多组织类型中一致的高灵敏度和空间分辨率。
为了验证MAGIC-seq的性能,研究团队将其与其他主流空间转录组技术进行了系统对比,包括商业平台10x Visium V2、微流体技术DBiT-seq和Decoder-seq等。结果显示,MAGIC-seq在多项关键性能指标上表现出显著优势:
基因和UMI检测数量
在小鼠脑切片实验中,MAGIC-seq在50 µm采集点处检测到的中位UMI数量为15,563个,检测到的基因数量为5576个,相比其他方法显著提高。例如,DBiT-seq在相同条件下的UMI数量仅为10,000个左右。这表明MAGIC-seq具有更高的基因检测能力,能够在低深度测序下获得更多的基因信息。
高通量与低成本的优势
MAGIC-seq在采集面积、采集点数量和成本上也表现出优势。在50 μm分辨率下,MAGIC-seq的采集面积达到21.6 mm × 21.6 mm,包含202,500个采集点,而DBiT-seq、10x Visium和Decoder-seq的采集点数量分别仅为2500、44,100和10,000个。MAGIC-seq在同等条件下,每平方毫米的成本仅为0.11美元,相比10x Visium和Decoder-seq分别降低了89%和50%以上。这种高效的设计使得MAGIC-seq在大规模样本处理上具有明显的成本优势。
基因表达一致性与重复性
MAGIC-seq在检测不同小鼠脑切片时表现出极高的重复性。在对同一玻片上的多个切片进行基因表达相关性分析时,发现相关系数均在0.997以上。这表明,MAGIC-seq能够有效地消除不同实验批次之间的差异,显著降低了批次效应的影响。
不同组织类型的高灵敏度检测
MAGIC-seq被应用于九种不同的小鼠组织(包括嗅球、脑、小肠、胎盘、肺、心脏、肝脏、脾脏和肾脏),结果表明每个采集点平均检测到5900多个基因。尤其是在嗅球和小脑的采集点中,检测到的基因数量高达8000多个,展示了MAGIC-seq在检测不同组织时的高灵敏度。此外,即使在同一玻片上处理不同渗透时间的组织,MAGIC-seq也能保持良好的检测结果。
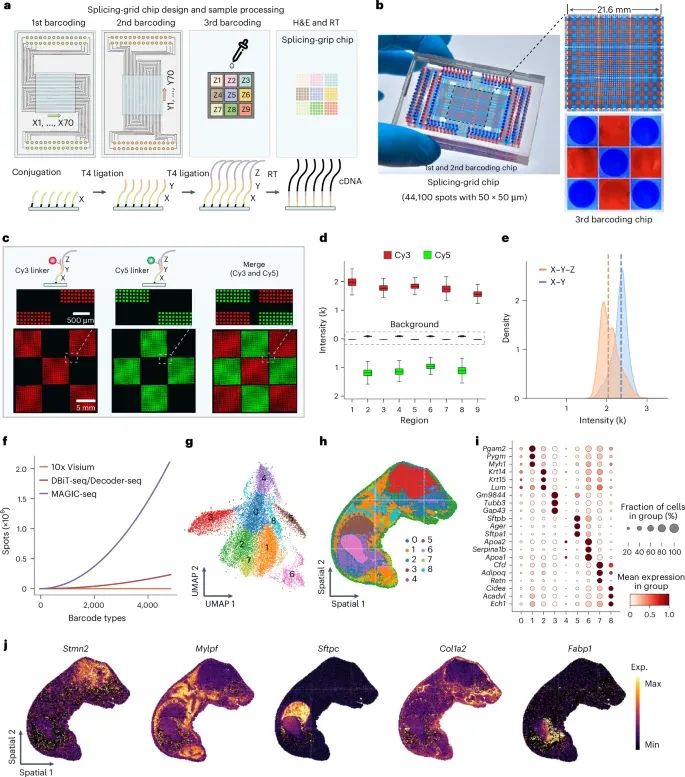
MAGIC-seq技术在大视野下对P3阶段小鼠进行空间映射的结果(Credit: Nature Genetics)
拼接网格芯片的设计与样本制备:图a概述了拼接网格芯片的设计过程,展示了如何通过三个九方格芯片实现更大的采集面积。通过H&E染色和渗透处理,样本的mRNA被捕获并进行逆转录,生成了空间转录组数据。
拼接网格芯片的制造过程:图b展示了用来制造拼接网格芯片的微流体装置。每个九方格芯片具有70个宽度为50 µm的通道,形成9个子网格。通过将多个子网格拼接在一起,最终实现了一个面积为21.6 mm × 21.6 mm的拼接网格,这显著扩大了捕获面积。
液体泄漏评估:图c和图d展示了拼接网格芯片中不同子网格之间的液体泄漏测试,结果显示,Cy3和Cy5荧光标记物在相邻网格中没有发生混合,表明子网格之间的分离有效,条形码的特异性得到了保持。
荧光强度和条形码效率:图e展示了两轮条形码标记(X–Y)和三轮条形码标记(X–Y–Z)的荧光强度分布,三轮条形码的效率约为87%,这表明在扩大捕获面积的同时,条形码标记的效率依然较高。
条形码效率比较:图f对比了MAGIC-seq与其他空间转录组方法(如10x Visium、Decoder-seq和DBiT-seq)的条形码编码效率,MAGIC-seq的条形码编码效率显著高于其他方法,这使其能够在更大面积上实现高效的空间转录组分析。
P3小鼠的空间映射:图g和图h展示了使用MAGIC-seq对P3阶段小鼠进行的空间基因表达映射,展示了不同组织(如大脑、肺、消化道、脂肪组织等)的基因表达集群。这些集群显示了良好的连续性和空间一致性,即使在相邻网格分离的情况下,相关基因的表达模式依然保持一致。
特征基因的空间分布:图i和图j展示了选定的标志性基因在P3小鼠组织中的空间分布,进一步验证了MAGIC-seq在大视野空间映射中的高分辨率和准确性。
3D空间转录组图谱的构建
MAGIC-seq不仅能够对二维空间样本进行高效分析,还能够用于构建大视野的三维(3D)空间转录组图谱。研究团队成功生成了一个小鼠发育中脑的3D转录组图谱。通过分析发育中小鼠大脑不同区域的93个切片,研究人员生成了总计98,192个采集点的三维分子图谱,每个采集点中检测到的基因数量中位数为4,880个。这种高分辨率的3D图谱为科学家提供了全新的视角,帮助深入了解小鼠大脑发育过程中不同细胞类型的空间分布。
通过MAGIC-seq生成的3D图谱,研究人员能够清晰地观察到兴奋性神经元(Glutamatergic Neurons)和抑制性神经元(GABAergic Neurons)在小鼠大脑中的空间分布差异。例如,在背侧皮层(Dorsal Pallium)和中央皮层(Medial Pallium)区域,兴奋性神经元占据主要比例,而在小脑核团和丘脑区域,抑制性神经元则更为丰富。此外,研究还发现,非神经元细胞如胶质细胞在脑膜区域中占据重要位置。
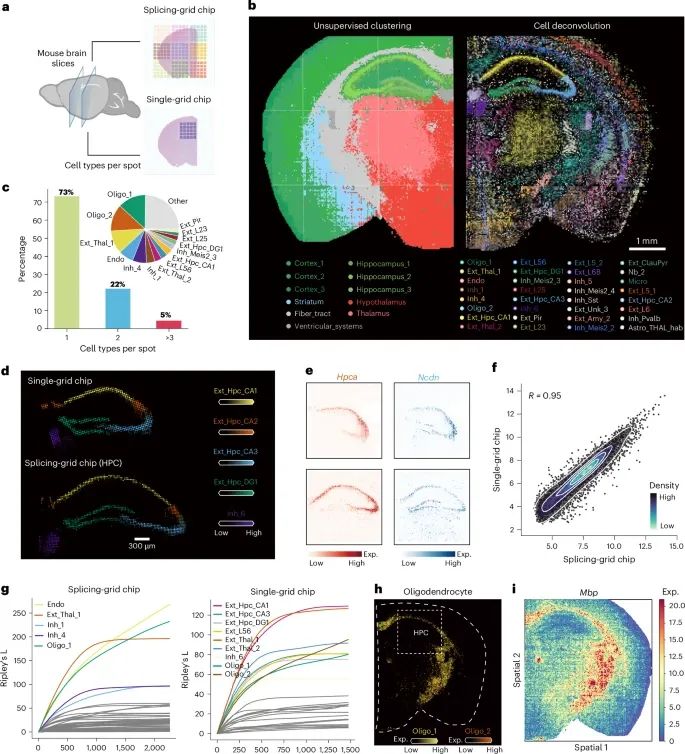
MAGIC-seq在细胞分辨率下对小鼠大脑进行空间映射的结果(Credit: Nature Genetics)
细胞分辨率空间映射概述:图a展示了MAGIC-seq在15 µm分辨率下对小鼠大脑进行空间分析的工作流程。研究人员通过拼接网格芯片覆盖整个半个大脑的区域,而单网格芯片仅能覆盖部分海马区域。这说明MAGIC-seq能够在更大视野下进行高分辨率的空间基因表达分析。
细胞类型无监督聚类与空间映射:图b展示了MAGIC-seq对小鼠大脑半球进行的无监督聚类结果,左侧展示了不同细胞类型的聚类图,右侧展示了这些细胞类型在大脑不同区域的空间分布。白色虚线代表了拼接网格芯片的分隔线,显示了基因表达在大脑结构中的高精度再现。
细胞类型数目的分布:图c展示了每个采集点包含的细胞类型数量分布。大部分采集点仅包含一种细胞类型(约73%),有些点包含两种细胞类型(约22%)。这一结果表明,MAGIC-seq具有较高的分辨率,能够在接近单细胞水平上准确区分不同的细胞类型。
海马区神经元类型的空间分布:图d展示了单网格芯片和拼接网格芯片在海马区不同类型神经元的空间分布图。拼接网格芯片的结果显示了更为精细的海马区神经元分布,具体包括海马角(CA1、CA2、CA3)和齿状回(DG)区域的不同神经元类型。
海马区标志基因的空间表达:图e展示了海马区标志基因在单网格芯片和拼接网格芯片中的空间分布情况。拼接网格芯片能够覆盖整个海马区域,而单网格芯片仅能捕捉到部分区域的基因表达,说明拼接网格芯片在大视野下能够提供更全面的基因表达信息。
海马区基因表达的相关性分析:图f展示了单网格芯片与拼接网格芯片在海马区基因表达上的相关性分析,结果显示两个芯片之间的基因表达模式具有高度相关性,证明了MAGIC-seq在不同芯片条件下能够保持一致的基因表达结果。
细胞聚类模式分析:图g通过Ripley’s L统计方法分析了两种芯片中细胞类型的聚集模式。结果显示,髓鞘细胞(Mbp标志基因)的聚集模式在拼接网格芯片中更加明显,而在单网格芯片中由于采集面积的限制,聚集模式较为分散。
髓鞘细胞的空间分布:图h和图i展示了髓鞘细胞在小鼠大脑中的空间分布情况,拼接网格芯片显示了髓鞘细胞在大脑中更为连续的分布,尤其是在海马区域的分布更加详细和完整。
为了进一步验证MAGIC-seq的适用性,研究团队还生成了小鼠发育过程的时空转录组图谱,覆盖了从E17.5到P4的关键发育阶段。通过对小鼠的肺、肝、心脏等器官的分析,MAGIC-seq揭示了这些器官在发育过程中基因表达的动态变化。例如,在P0和P4阶段,小鼠肺部的表面活性物质基因(Sftpc, Sftpb)的表达显著增加,这对于新生儿的肺功能至关重要。而在小鼠肝脏中,研究团队检测到了肝母细胞标志物(如Afp)的持续高表达,表明出生前后肝脏细胞仍在进行活跃的增殖。
通过对小脑发育的精细分析,研究团队发现小脑皮层的分层结构逐渐在E17.5到P4阶段形成,包括外颗粒层(External Granular Layer)、Purkinje细胞层(Purkinje Cell Layer)和内颗粒层(Internal Granular Layer),并且这些区域的基因表达表现出显著差异。
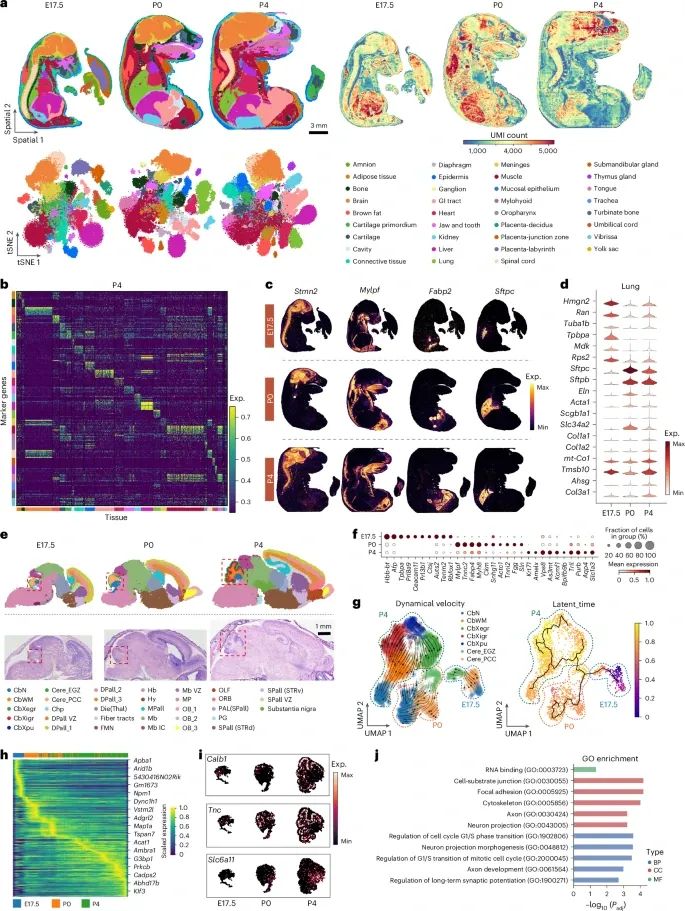
MAGIC-seq技术用于小鼠器官发生时空转录组图谱的构建(Credit: Nature Genetics)
小鼠器官的发育时间点:图a展示了P0到P4阶段不同小鼠器官的发育,包括肺、肝、肾、脾、心脏和大脑等。通过MAGIC-seq技术,研究人员能够在不同发育阶段捕捉这些器官的空间基因表达动态变化。每个器官的切片分别进行了时空转录组测序,绘制了基因表达的空间分布图。
肺组织的空间表达动态:图b展示了小鼠肺在P0到P4阶段的发育变化,特别是与表面活性物质合成相关的基因(如Sftpc和Sftpb)的空间分布。结果显示,这些基因在P0到P4阶段的表达显著增加,表明新生儿肺的表面活性物质合成对于肺功能的正常发育至关重要。
肝脏的空间转录组图谱:图c展示了小鼠肝脏在不同发育阶段的基因表达情况,尤其是与肝细胞增殖相关的标志基因(如Afp)的空间分布。研究显示,肝脏在出生前后仍保持活跃的细胞增殖状态,这一过程对肝脏的成熟和功能完善具有重要作用。
肾脏的基因表达分析:图d展示了肾脏中不同基因在不同发育阶段的空间表达情况。特定标志基因(如Wnt4)的表达揭示了肾脏在出生前后发育中的重要调控过程。MAGIC-seq提供的高分辨率空间转录组数据有助于理解肾脏组织的功能性分区和细胞类型特异性。
心脏发育的基因表达:图e展示了心脏发育过程中心肌细胞和内皮细胞的基因表达情况。心脏发育中的细胞类型及其空间分布,尤其是与血管生成相关的基因(如Pecam1)的表达,显示了MAGIC-seq技术在解析心脏组织空间结构中的有效性。
脾脏和大脑的发育图谱:图f,g分别展示了脾脏和大脑在不同发育阶段的基因表达分布,揭示了器官特异性基因在发育过程中空间表达的变化。例如,脾脏中与免疫功能相关的基因表达随着发育进程而增强,而大脑中神经元类型的分布和动态变化则与大脑的结构发育密切相关。
不同器官发育中的基因表达动态分析:图h通过热图展示了不同发育阶段、不同器官中特定基因的时空表达变化。每个器官的发育都伴随着一组特定基因的显著变化,这些基因的表达模式为理解器官发生的分子机制提供了宝贵的线索。
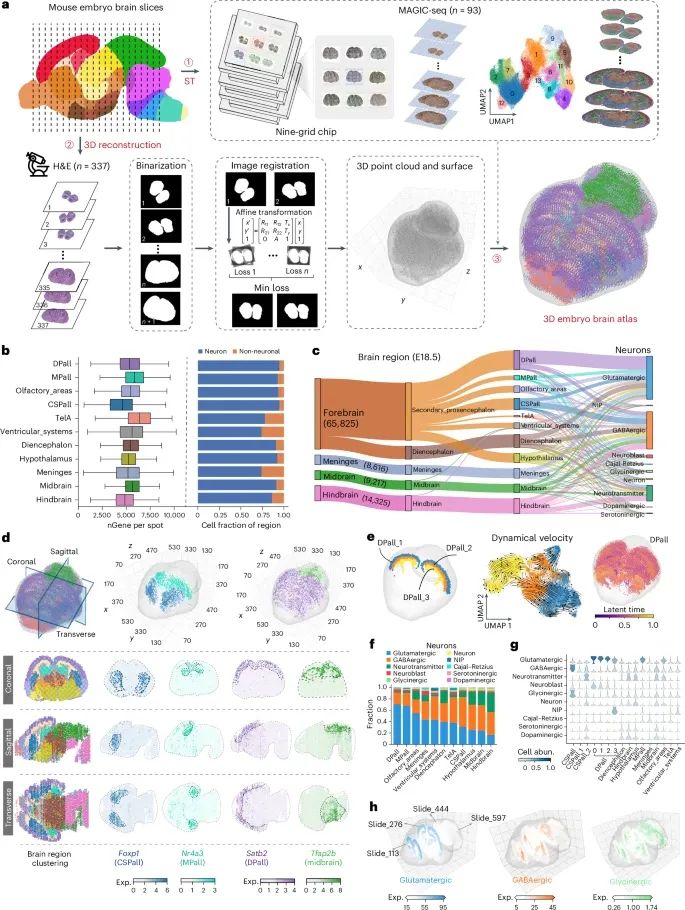
MAGIC-seq技术对E18.5阶段小鼠大脑进行三维(3D)转录组图谱构建的结果(Credit: Nature Genetics)
E18.5小鼠大脑的3D转录组图谱构建:图a展示了使用MAGIC-seq对E18.5阶段小鼠大脑进行了93个切片的空间转录组测序,生成了大脑的三维转录组图谱。每个切片中都进行了高分辨率的空间基因表达分析,覆盖了大脑的各个区域。研究人员将这些切片的数据整合为一个完整的3D图谱,能够精确地展现基因表达在大脑中的空间分布。
小鼠大脑不同区域的基因表达聚类:图b展示了通过无监督聚类分析对大脑不同区域进行的细胞类型分类。研究人员将这些聚类结果映射到3D空间中,揭示了小鼠大脑各个区域中不同细胞类型的空间分布。例如,皮层、丘脑和小脑区域的细胞类型有着清晰的空间界限,这种基因表达的空间异质性为研究大脑区域的功能提供了分子依据。
小鼠大脑的空间基因表达图谱:图c展示了大脑中多个标志性基因的空间表达模式。通过对这些基因的分析,研究人员能够揭示大脑中不同功能区域的基因表达差异。例如,皮层中的兴奋性神经元标志基因(Slc17a7)和抑制性神经元标志基因(Gad1)在不同的大脑区域有着特异性表达。这些空间表达数据帮助科学家进一步了解大脑各个功能区的基因表达特征。
皮层区域的基因表达详细分析:图d展示了皮层不同层次中基因表达的空间分布,特别是层特异性基因(如Reln、Bcl11b、Tbr1)在皮层中的表达模式。这些基因在皮层的不同层次中表现出显著差异,说明大脑皮层已经在E18.5阶段形成了分层结构。图e和f进一步细化了这些层特异性基因的表达情况,揭示了皮层发育中的复杂分层模式。
小脑区域的基因表达特征:图g展示了小脑中神经元类型和标志基因的空间表达模式。研究显示,E18.5阶段的小脑已经具有明显的分区结构,特定神经元类型(如Purkinje细胞)在小脑的不同区域中表现出特异性的空间分布。
基因表达的三维映射与整合:图h展示了通过将所有切片的数据整合到三维坐标系中,生成的E18.5小鼠大脑的三维基因表达图谱。该图谱能够在三维空间中清晰地展示不同基因在大脑各个区域的表达情况,从而帮助研究人员理解不同大脑区域的功能分化及其分子机制。
MAGIC-seq是一项创新的空间转录组技术,它通过其高通量、低成本和大视野的设计,成功解决了现有ST方法的诸多瓶颈。通过该技术,研究人员能够以极高的分辨率捕捉复杂组织的空间基因表达,并生成大规模的3D空间转录组图谱。MAGIC-seq的应用前景广泛,不仅在神经科学和发育生物学研究中发挥重要作用,还能够为多组学研究提供宝贵的技术支持。
参考文献
Zhu J, Pang K, Hu B, He R, Wang N, Jiang Z, Ji P, Zhao F. Custom microfluidic chip design enables cost-effective three-dimensional spatiotemporal transcriptomics with a wide field of view. Nat Genet. 2024 Sep 10. doi: 10.1038/s41588-024-01906-4. Epub ahead of print. PMID: 39256584.
https://www.nature.com/articles/s41588-024-01906-4


