Nat Commun:中科大瞿昆/郭闯团队对染色体外环状DNA算法性能进行系统评估
时间:2024-11-15 18:02:53 热度:37.1℃ 作者:网络
染色体外环状DNA(eccDNA)通常携带关键的致癌基因和调控元件(如启动子和增强子),在肿瘤研究中具有重要意义。随着研究的深入,人们对eccDNA及其在癌基因扩增、基因表达调控、基因组重排和肿瘤内异质性中作用的理解也逐步加深。目前已有多种分析算法和实验方法来检测eccDNA,包括AmpliconArchitect(AA)算法、CReSIL、Circle_finder、Circle-seq和3SEP等。但鉴于eccDNA结构的复杂性及大小的多样性,针对不同研究选择最适合的分析算法和实验方法仍是一项复杂的任务。现有eccDNA检测方法的评估通常范围有限,往往集中在精确度或计算需求等单一方面,并依赖于过于简化的模拟,无法代表真实测序数据的复杂性。
为解决上述难题,中国科学技术大学瞿昆、郭闯团队在Nature Communications发表了题为“Comparative analysis ofmethodologies for detecting extrachromosomal circular DNA”的文章。研究团队使用7个模拟数据集分析了7种在测序数据中鉴定eccDNA的算法,评估其在准确性、身份识别、重复率和计算资源消耗方面的性能;并通过21个真实测序数据集比较了7种实验建库方法对不同eccDNA类型的检测效率。该比较研究突出了分析富含ccDNA的短读长和长读长测序数据最有效的方法,强调了不同实验方法中eccDNA检测效率的差异,有助于指导科研人员选择合适的研究方法,促进高效eccDNA检测新方法的开发。

文章发表在Nature Communications
01 研究设计
为评估不同分析流程在eccDNA识别中的性能,研究团队开发了一个Python脚本生成eccDNA模拟数据集。该脚本从现有数据中推断长度分布、染色体起源和嵌合的eccDNA比例,以创建模拟环状DNA(真阳性)和线性DNA(真阴性)的混合数据,并模拟了滚环扩增(RCA)过程。最终生成了7个模拟数据集,每个数据集在50X深度下包含10,000个环状和10,000个线性DNA序列。
研究团队评估了7种算法的11种模式,包括用于短读长(SR)测序数据分析的Circle-Map、Circle_finder(bwa-mem-samblaster和microDNA.InOne.sh)、ECCs_plorer和ecc_finder(map-sr和asm-sr),以及用于长读长(LR)测序数据分析的CReSIL、eccDNA_RCA_nanopore、NanoCircle和ecc_finder(map-ont和asm-ont)。性能指标包括F1评分和鉴定的eccDNA与模拟的eccDNA之间的碱基对差异。
对于实验方法评估,研究团队选择了Circle-Seq(SR和LR)、3SEP(SR和LR)、WGS(SR和LR)和ATAC-Seq(SR),评估了每种方法在不同长度和拷贝数状态下的eccDNA检测效率。
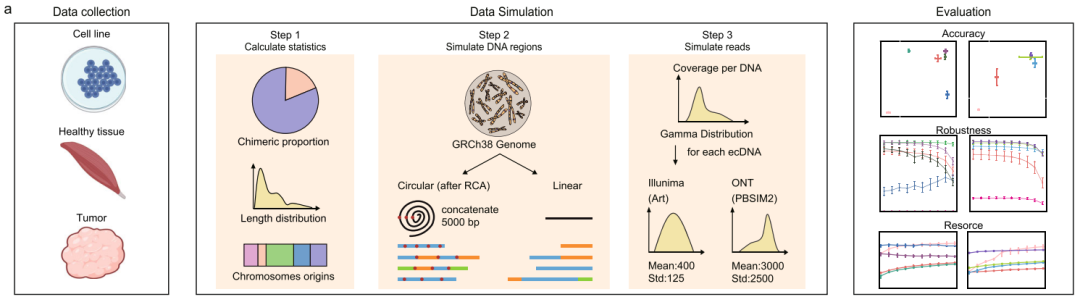
图1. 实验流程
02 eccDNA鉴定中不同分析算法的评估
在模拟测序深度为50×时,研究团队对每种分析算法在eccDNA鉴定中的性能进行了评估。结果显示,Circle_finder(bwa-mem-samblaster)和Circle-Map在短读长测序数据中的表现优于其他方法,F1评分分别达到0.912和0.908;CReSIL在长读长测序数据中表现最佳,F1评分为0.918、碱基对差异为4.160 bp。
接下来,研究团队将模拟数据集设置为不同的测序深度,评估了每种算法在eccDNA鉴定中的性能。对于短读长测序数据,Circle_finder(bwa-mem-samblaster)、Circle-Map在所有测序深度中F1评分始终最高;当测序深度从50×下降到5×时,Circle-Map和Circle_finder(microDNA.InOne.sh)的碱基对差异保持稳定;ecc_finder在所有测序深度中显示最低F1评分。在长读长测序数据中,CReSIL在深度超过10×时具有最高的F1评分,而eccDNA_RCA_nanopore在深度低于10×时表现出优越的性能。
除测序深度外,研究团队还研究了嵌合DNA对eccDNA鉴定性能的影响。对于短读长测序数据分析,嵌合DNA比例的变化不影响Circle-finder(bwa-memsamblaster)、Circle-Map和ecc_finder(map-sr)的eccDNA鉴定召回率,但影响ECCs_plorer。在长读长测序数据分析中,对于简单eccDNA和嵌合的eccDNA鉴定,大多数算法基本保持一致的召回率。
基于上述分析,Circle_finder(bwa-memsamblaster)和Circle-Map是最合适分析富集eccDNA的短读长测序数据的算法,但Circle_finder容易生成冗余结果;CReSIL在分析富集eccDNA的长读长测序数据方面优于其他算法,其检测准确率较高,碱基对差异较小。
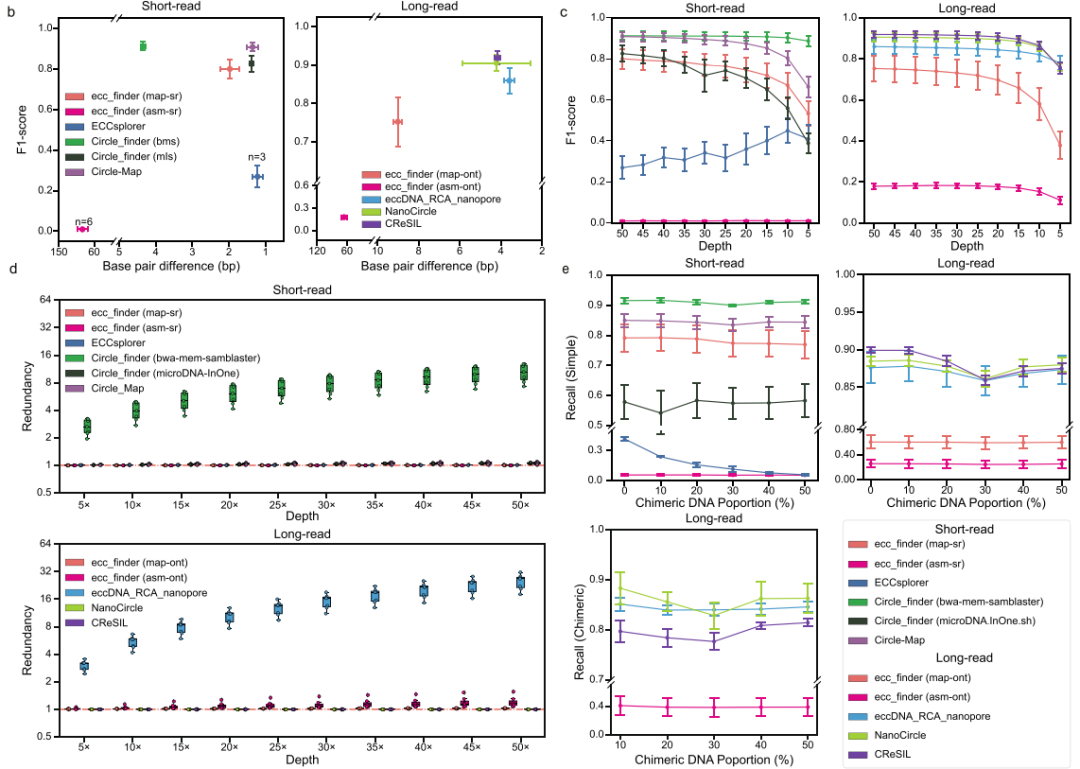
图2. eccDNA鉴定中不同分析算法的评估
03 富集步骤对eccDNA鉴定的影响
接下来,研究团队通过每Gb数据检测到的eccDNA数量来评估eccDNA检测效率。结果显示,与不采用RCA相比,采用RCA步骤的方法eccDNA检测效率显著更高;基因组拷贝数与重叠eccDNA的覆盖率之间存在正相关。
对eccDNA长度分布和染色质来源的进一步分析表明,富集方法检测的eccDNA有97%以上短于10kb,而非富集方法检测到的eccDNA长度超过10kb的比例较高。除3SEP-SR和WGS-SR外,大多数方法的eccDNA密度(每百万碱基检测到的eccDNA数量)与染色体上的蛋白质编码基因密度之间呈显著正相关。
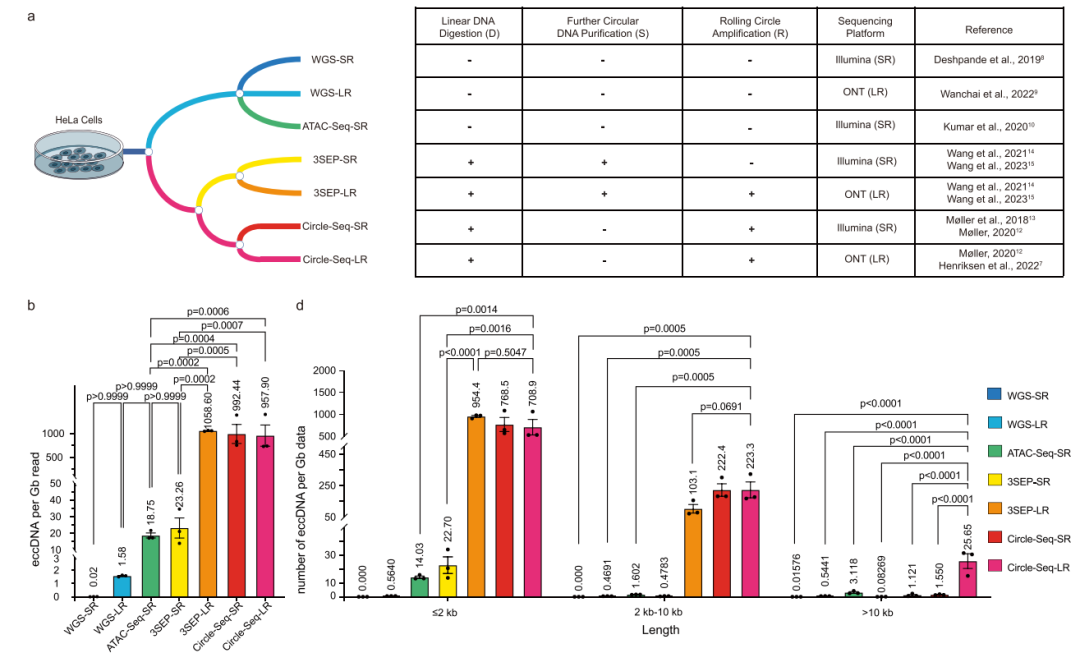
图3. eccDNA富集操作对eccDNA鉴定的影响
04 不同实验方法对ecDNA的检测效率
与拷贝数扩增区重叠的eccDNA被归类为ecDNA,而这些区域之外的ecDNA被归类为非ecDNA。研究发现,Circle-Seq-SR、Circle-Seq-LR和3SEP-LR在每Gb数据中鉴定出的ecDNA平均数量较高,但WGS-SR、WGS-LR和ATACSeq-SR鉴定出的eccDNA中ecDNA的比例明显更高。
研究团队进一步分析了不同长度(≤2kb,2-10kb,>10kb)ecDNA和非ecDNA的检测效率。结果显示,3SEP-LR在检测长度≤2kb的ecDNA和非ecDNA时显示出最高的效率;Circle-SeqSR对2-10kb的ecDNA检测效率最高;对于>10kb的ecDNA, Circle-Seq-LR检测性能优于其他方法。
此外,不同实验方法检测到的ccDNA图谱具有异质性,其检测到的eccDNA在长度、癌基因组成和包含的基因重复元件等方面展现出显著不同。因此,在比较不同研究的结果时,特别需要考虑所使用的实验方法。
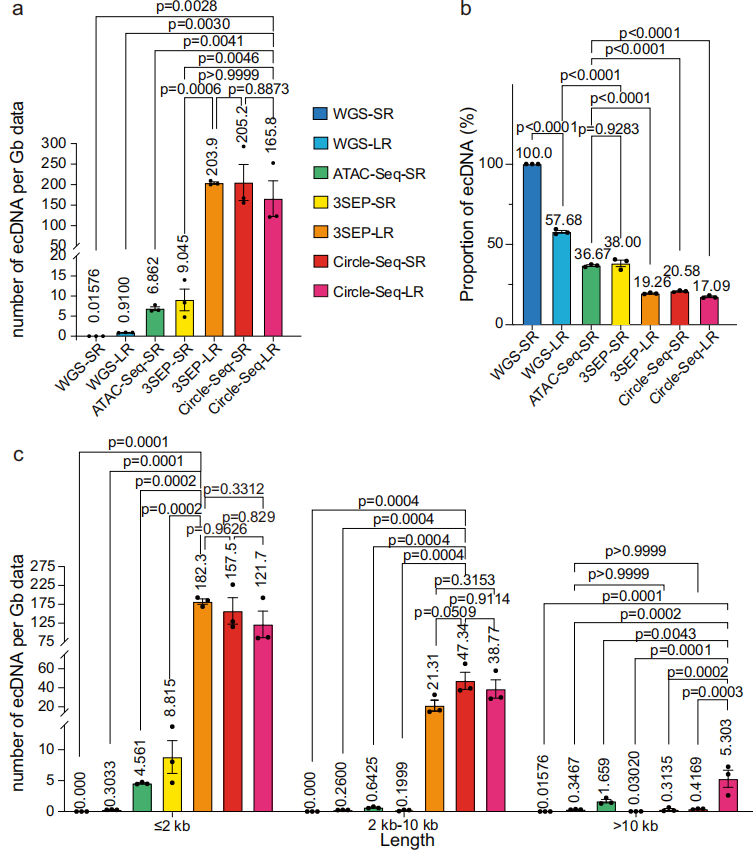
图4. 7种实验方法对ecDNA的检测效率
05 结 语
综上所述,研究团队使用各种指标评估了7种分析算法、并通过检测效率比较了7种实验方法,确定了最佳eccDNA检测方法。Circle_finder(bwamem-samblaster)和Circle-Map在短读长数据中鉴定eccDNA的性能最佳,而CReSIL在长读长数据分析方面表现优于其他方法。在实验方法中,Circle-Seq-LR对较长的eccDNA检测效率最高,而3SEP-LR对较短的eccDNA的检测效率更高。该研究结果为科研人员选择最合适的eccDNA研究方法提供了重要信息。
论文原文:
Gao, X., Liu, K., Luo, S. et al. Comparative analysis of methodologies for detecting extrachromosomal circular DNA. Nat Commun 15, 9208 (2024).
https://www.nature.com/articles/s41467-024-53496-8


