Science:中山大学舒跃龙团队破解H7N9禽流感感染之谜
时间:2021-08-21 20:02:58 热度:37.1℃ 作者:网络
流感病毒种类多,宿主范围广,广泛分布在野鸟、水禽、陆禽等多种自然宿主中,近年来,越来越多禽流感病毒跨宿主传播感染人类,严重危害人类健康,同时给全球公共卫生安全带来重大挑战,目前,可以感染人类的禽流感病毒主要包括H5N1、H5N6、H7N9以及H9N2等。这些病毒不断跨种传播的现象提醒我们,再次发生流感大流行的威胁始终存在,世界卫生组织也一直将流感大流行作为人类面临的十大公共卫生威胁之一。
2013年春,中山大学舒跃龙教授团队在国际上首次报道一种新型重配低致病性H7N9禽流感病毒(Low pathogenic avian influenza A (H7N9) virus,LPAI H7N9)可导致人的感染和死亡(NEJM 2013)。H7N9禽流感病毒在人群中共造成了五波流行,导致我国内地1537人感染,其中612例死亡,病死率约39.8%。流行病学研究证明活禽市场和家禽暴露是感染H7N9禽流感病毒的主要风险因素,但H7N9禽流感病例中,职业暴露人群(与家禽养殖、运输以及销售等有关的从业人员)只占总确诊人数的7%。
那么,相同暴露条件下,为何只有很少的人会感染H7N9禽流感病毒,而大部分人则不被感染,仍是未解之谜。
为了攻克这个难题,舒跃龙教授团队推测宿主遗传因素可能发挥了重要作用。为了证明这个假设,团队采用病例对照研究方法,共纳入217例H7N9禽流感确诊病例(real time RT-PCR检测阳性)和116例与确诊病例具有流行病学关联的职业暴露人群对照(real time RT-PCR检测阴性),收集样本并进行人全基因组测序。通过全基因组关联分析和Gene-base关联分析,首次发现人MX1基因稀有变异可能增加人感染H7N9禽流感病毒风险。
该研究以:Rare variant MX1 alleles increase human susceptibility to zoonotic H7N9 influenza virus 为题,于2021年8月20日在线发表于 Science 期刊。

抗粘液病毒基因(Myxovirus resistant, Mx)是最早发现的干扰素诱导的抗病毒基因之一,人MX1基因可编码人抗黏液病毒蛋白A(Human myxovirus resistance protein A, MxA),MxA蛋白属于动态蛋白超家族的一类大分子GTPase,对多种RNA病毒和DNA病毒具有抗病毒作用,包括甲型流感病毒和乙型肝炎病毒等。
进一步的研究结果表明,H7N9禽流感病例中共发现了17个MX1稀有变异,包括15个错义突变、1个无义突变和1个剪切位点突变。体外实验验证表明,其中14个变异会影响蛋白功能。流感聚合酶活性实验及病毒感染实验表明,这14个携带突变的MxA蛋白不仅失去对H7N9禽流感病毒的抑制作用,也失去对H7N7和H5N1禽流感病毒的抑制作用,同时也失去对pdmH1N1流感的抑制作用。
经过一代测序验证,H7N9病例携带的MX1 稀有突变全部为杂合突变,那么野生型MxA是否在体内继续发挥抗病毒作用呢?
研究团队进一步开展了MxA突变体对野生型MxA的显性负效应研究,结果表明有12个失去抗病毒活性的MxA突变体对野生型MxA具有显性负效应,说明MX1杂合子不受保护。这项研究首次发现与人感染H7N9禽流感病毒风险增加相关的MX1基因变异,进一步通过体外实验验证,MX1基因稀有变异可以增加人感染禽流感病毒风险。
研究结果首次解释了相同暴露条件下,为何只有一小部分人会感染H7N9禽流感病毒这一流行病学现象,同时这些变异均为稀有变异,在人群中的变异频率小于0.5%,间接提示H7N9禽流感病毒目前导致流感大流行的风险较低;研究结果也为将来可能的高危人群监测提供了科学依据,进一步推动传染病精准防控。
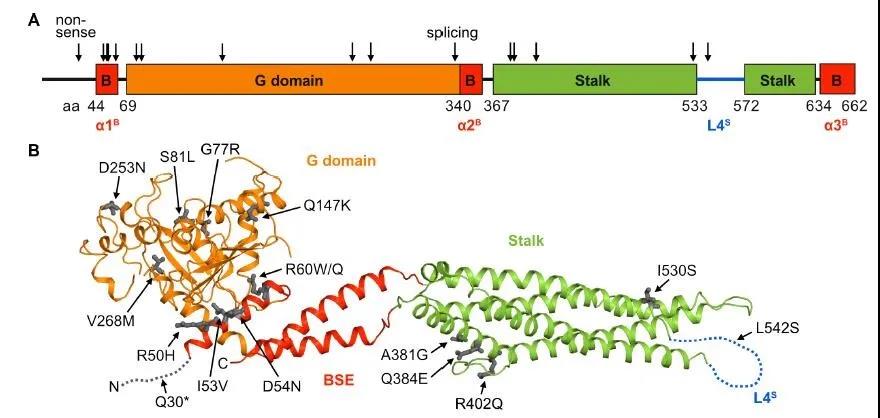
H7N9禽流感病例中发现的MX1稀有变异示意图
中山大学公共卫生学院(深圳)陈永坤博士、德国弗莱堡大学博士后Laura Graf、中山大学公共卫生学院(深圳)博士研究生廖启军和中国疾病预防控制中心病毒病预防控制所陈涛副研究员为论文共同第一作者,中山大学公共卫生学院(深圳)舒跃龙教授、德国弗莱堡大学Martin Schwemmle教授和中国疾病预防控制中心病毒病预防控制所王大燕研究员为共同通讯作者。本研究得到国家重点研发计划和深圳市科技计划项目的支持。
原始出处:
Yongkun Chen, et al. Rare variant MX1 alleles increase human susceptibility to zoonotic H7N9 influenza virus. Science 20 Aug 2021: Vol. 373, Issue 6557, pp. 918-922. DOI: 10.1126/science.abg5953.


