Cell:AI重大突破!发现7万种全新病毒,新工具揭秘病毒“暗物质”
时间:2024-10-15 06:02:12 热度:37.1℃ 作者:网络
在刚刚颁布的诺贝尔自然科学奖项中,人工智能(AI)成了引发全球热议的最大“赢家”。其中,诺贝尔物理学奖出人意料地授予了“AI教父”Geoffrey Hinton博士与John Hopfield教授,以表彰他们在机器学习领域的开创性贡献;而“AlphaFold之父”Demis Hassabis博士和John Jumper博士,以及华盛顿大学David Baker教授则是借助AI工具,分别因蛋白质结构预测和计算蛋白设计的贡献获得诺贝尔化学奖。
事实上,不止是蛋白结构预测与设计,AI已经对生命科学研究带来了全方位的影响。就在本周,一项发表于《细胞》的研究再次展示了AI的强大能力。来自中山大学医学院、阿里云与悉尼大学的研究团队利用其开发的深度学习算法LucaProt,发现了超过16万种RNA病毒(包括7万种首次发现的新病毒),揭示了大量前所未知的病毒“暗物质”。这项研究大幅拓展了人类对病毒界的认知,并为记录全球RNA病毒组提供了全新的工具。

RNA病毒不仅是感染各种宿主、导致人类疾病的罪魁祸首,也是在全球生态系统中无处不在、发挥关键作用的组成部分。近些年的研究通过对RNA依赖性RNA聚合酶(RdRP)序列的分析,已经鉴定出了数万种新病毒。
虽然科学家们通过生态采样和测序,在RNA病毒多样性方面已经取得了巨大进展,但这些研究也受到了限制——它们依赖于已知病毒序列同源性,因此对于经过高度分化、无法通过序列同源性识别的病毒“暗物质”,科学界仍然无能为力。
已有研究利用深度学习方法,从宏转录组等数据中识别病毒。这些工具采用了卷积神经网络(CNN)和循环神经网络(RNN)。不过,CNN和RNN在处理生物序列时都面临限制:前者存在捕捉全局相关性的挑战,后者则难以处理较长的序列。
在最新研究中,研究团队转而采用了transformer架构。transformer架构能有效地适应不同长度的序列,并且捕获序列位置之间的局部和长距离关系,具有超越CNN和RNN的能力。
基于这些优势,研究团队利用transformer架构设计了识别高度分化的RNA病毒的工具——LucaProt。该工具整合了蛋白质序列信息以及病毒RdRP序列的结构特征,运用深度学习方法对全球不同生态系统中的10487份宏转录组数据进行挖掘。
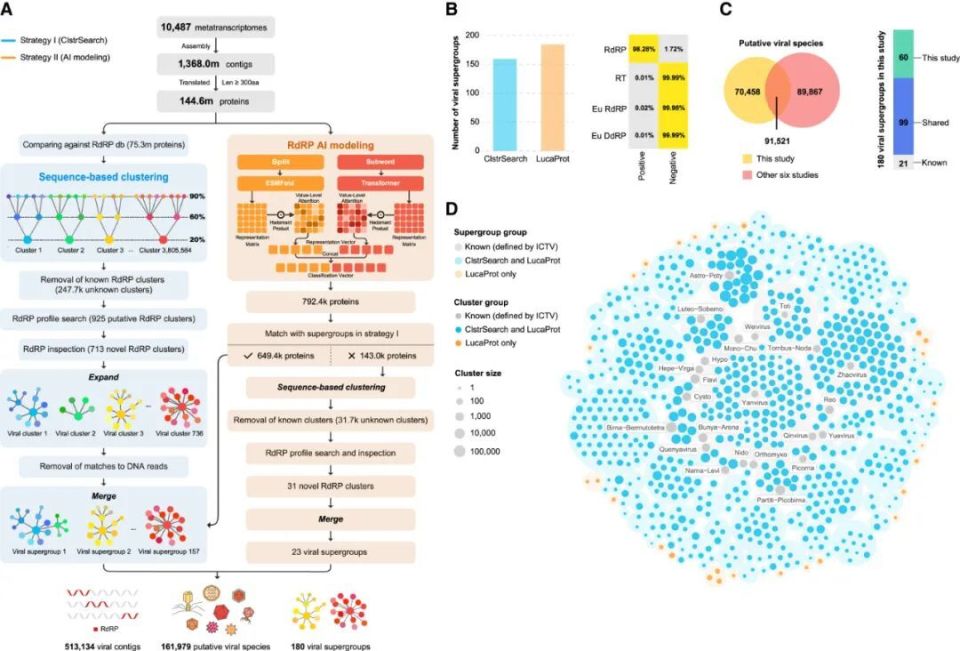
▲研究团队基于该流程发掘RNA病毒,并且发现了23个此前无法识别的病毒超群(图片来源:参考资料[1])
结果,研究发现了161979个潜在的RNA病毒物种,它们涵盖了180个RNA病毒超群(在分类学中相当于门或纲)。其中,有70458种是由LucaProt首次发现的RNA病毒新物种。值得一提的是,在180个RNA病毒超群中,有23个属于此前无法识别的病毒“暗物质”。
这些病毒分布在各种极端的生态系统中,包括海底热液、超盐湖泊、盐沼等。其中,这项研究还找到了包含47250个核苷酸的已知最大RNA病毒,展示出极高的遗传物质复杂性。由此,这项研究揭示了前所未有的病毒多样性,大幅拓展了对病毒圈的认知。
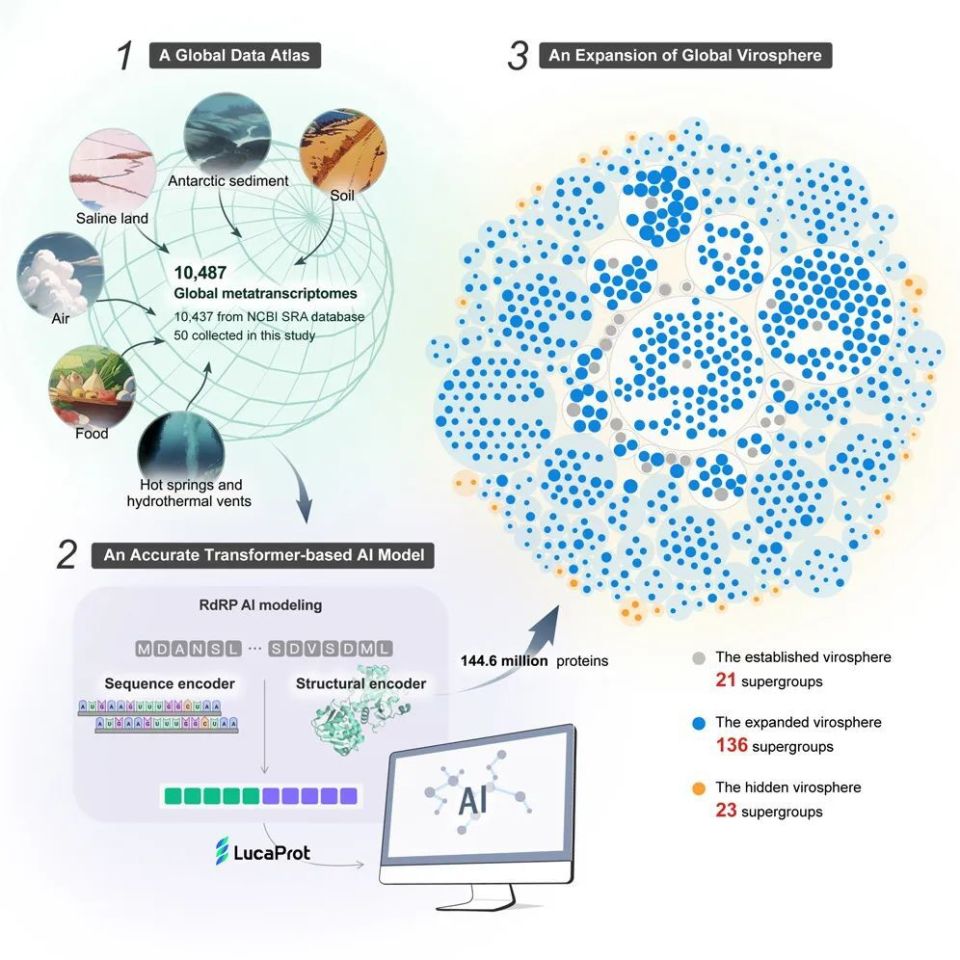
▲研究示意图(图片来源:参考资料[1])
综上,这项研究通过AI与病毒学的融合,展示了AI在进行生命科学探索中的高效性。这种学科交叉为进一步解析生命序列、拓展对生态系统的认识提供了宝贵的见解。
参考资料:
[1] Xin Hou et al., Using artificial intelligence to document the hidden RNA virosphere. Cell, 2024; DOI: 10.1016/j.cell.2024.09.027


